


Nos dias 12 a 15 de Setembro de 2022 aconteceu a 67ÂŞ edição do Congresso Brasileiro de GenĂŠtica, na cidade de Natal/RN, e o LabCECA foi representado pela discente de iniciação cientĂfica JĂşlia Tomaz. Na oportunidade a discente apresentou o trabalho intitulado “DIRS-like and Ngaro-like retrotransposons illuminate chromosomal diversification into Pipidae genomes”.Â
O trabalho apresentou dados inĂŠditos sobre a distribuição cromossĂ´mica de cĂłpias de elemento genĂŠtico mĂłveis das subfamĂlias DIRS-like e Ngaro-like.
Os discentes Pedro Macedo e Thiago Algusto Matsunga, ambos do curso de Ciências Biológicas, apresentaram parte dos resultados obtidos em seus projetos de PIBIC, desenvolvidos no LabCECA, durante a IIIª Escola Paranaense de Bioinformåtica, na UFPR.
O evento reuniu pesquisadores nacionais que trabalham em projetos utilizando bioinformĂĄtica para resolução de questĂľes biolĂłgicas. No evento foram apresentadas novas abordagens metodolĂłgicas, pipelines e softwares destinados a anĂĄlises de dados em larga escala. Pedro trabalha com elementos genĂŠticos mĂłveis e seu papel na reorganização dos genomas e no evento apresentou dados sobre os elementos da famĂlia Keno e sua possĂvel predileção de inserção associada Ă genes de snoRNA da famĂlia U2 no genoma de Xenopus tropicalis. JĂĄ Thiago participa do projeto de pesquisa “Evolução de genes de receptores olfatĂłrios sobre um aspecto adaptativo” e apresentou a descrição do repertĂłrio de genes de receptores olfativo que compĂľe o genoma de duas espĂŠcies de anuros.
AlĂŠm do ineditismo de seus trabalhos em genĂ´mica estrutural de anfĂbios, estes trabalhos abrem novas perspectivas de estudos que serĂŁo agora ampliados em seus respectivos projetos. Certamente um Ăłtimo inĂcio em suas carreiras cientĂficas!
Os elementos transponĂveis (TEs) sĂŁo sequĂŞncias de DNA repetitivas e dispersas no genoma de organismos eucariotos. Possuem capacidade mĂłvel, podendo âsaltarâ de um local cromossĂ´mico para outro (caso estejam ativos) e por isso atuam como um importante substrato para a evolução dos genomas. Os chamados Retrotransposons se movimentam utilizando uma molĂŠcula de RNA intermediĂĄrio, cujo transcrito (RNA) ĂŠ utilizado para a sĂntese de uma nova cĂłpia de DNA, produzido atravĂŠs da atividade da enzima transcriptase reversa. Logo, a cada ciclo de transposição, esses elementos mantĂŠm uma cĂłpia em seu local de origem e inserem a nova cĂłpia em um novo local do genoma. Por essa razĂŁo, esses elementos estĂŁo diretamente relacionados com o aumento do tamanho dos genomas.
Sabendo da importância evolutiva desses elementos nos genomas, a biĂłloga Joana Moura Gama buscou investigar em seu mestrado qual a diversidade de retrotransposons e como esses elementos evoluĂram no genoma de trĂŞs espĂŠcies de sapos: duas espĂŠcies africanas (Xenopus tropicalis e Xenopus laevis) e uma espĂŠcie endĂŞmica do Brasil (Pipa carvalhoi). Essas espĂŠcies pertencem a uma mesma famĂlia de Anuros e sĂŁo proximamente aparentadas. Assim como as diferentes espĂŠcies sĂŁo catalogadas e recebem um ânome cientĂficoâ e sĂŁo ordenadas dentro de uma classificação taxonĂ´mica, TEs sĂŁo classificados em um sistema similar. Sob orientação do Prof. Daniel Bruschi (UFPR) e da Dra. Adriana Ludwig (ICC-Fiocruz), Joana trabalhou com retrotranspons da ordem LINE, que reĂşne diferentes famĂlias desses TEs. Cada famĂlia ĂŠ formada por grupos de sequĂŞncias que sĂŁo molecularmente semelhantes. Em seu mestrado, a discente encontrou uma diversidade de famĂlias de elementos comum entre as trĂŞs espĂŠcies (elementos das famĂlias Rex1, L2, Cr1, L1, TX1), que possivelmente foram herdadas a partir de um ancestral comum (estima-se que essas espĂŠcies partilharam de um Ăşltimo ancestral em comum a pelo menos 136 milhĂľes de anos!). Apenas uma famĂlia (RTE) nĂŁo esteve presente no genoma de Xenopus tropicalis, possivelmente tendo sido perdida durante a evolução desta espĂŠcie.
Os TEs sĂŁo transferidos verticalmente de uma espĂŠcie ancestral para as espĂŠcies derivadas durante o processo de especiação. Por isso dizemos que este tipo de transferĂŞncia segue o fluxo vertical da ĂĄrvore da vida, onde existe uma relação de parentesco entre as espĂŠcies que compartilham um mesmo TE. No entanto, algo foi bastante curioso entre os achados deste trabalho: dentro de algumas famĂlias de TEs foi observado um nĂvel surpreendente alto de similaridade (acima de 90%) entre as sequĂŞncias de TEs das espĂŠcies de anfĂbios estudadas e com sequĂŞncias de peixes. Esse dado foi inesperado, jĂĄ que peixes e anfĂbios partilharam de um Ăşltimo ancestral em comum a pelo menos 400 milhĂľes de anos. Esses achados contrariam as premissas da transferĂŞncia vertical, o que levou joana e seus colaboradores a testar uma hipĂłtese alternativa: a TransferĂŞncia Horizontal (TH) de sequĂŞncias de DNA. Este mecanismo tem sido utilizado para explicar a ocorrĂŞncia de sequĂŞncias de DNA idĂŞnticas (ou muito similares) no genoma de espĂŠcies que nĂŁo tem nenhuma proximidade filogenĂŠtica mas que ocupam o mesmo nicho ecolĂłgico. Curiosamente, as trĂŞs espĂŠcies de anfĂbios estudados tem seus adultos mantendo hĂĄbito de vida essencialmente aquĂĄtico, o que pode ter facilitado a troca de sequĂŞncias de DNA entre anfĂbios e peixes ao longo da evolução. Validaçþes laboratoriais precisam ainda ser realizadas para confirmar essa hipĂłtese, no entanto, estudos tem apontado inĂşmeros outros casos de TH em vertebrados, sendo os mais frequentes aqueles que envolvem a ocorrĂŞncia de TH entre espĂŠcies aquĂĄticas.
Os dados foram recentemente publicados no importante periódico internacional Molecular Phylogenetics and Evolution. A pesquisa contou com com a colaboração da Dra. Adriana Ludwig do Instituto Carlos Chagas, especialista em evolução de elementos genÊticos móveis, Dr. Dieval Guizelini do Programa de Pós-Graduação em Bioinformåtica na UFPR e da Dra. Shirlei Maria Recco-Pimentel do Departamento de Biologia Estrutural e Funcional da UNICAMP.
Ficou interessado em saber mais de TEs em anuros? Click no link e divirta-se.
As espĂŠcies Xenopus tropicalis e X. laevis sĂŁo dois importantes organismos-modelo para o estudo de evolução, genĂŠtica e biologia do desenvolvimento. As duas espĂŠcies estĂŁo distribuĂdas no continente africano e pertencem a famĂlia Pipidae. Desta mesma famĂlia, apenas o gĂŞnero Pipa ocorre na AmĂŠrica do Sul, sendo que no Brasil ocorrem cinco das sete espĂŠcies descritas para este gĂŞnero. Enquanto X. tropicalis apresenta 2n=20 cromossomos, a histĂłria de evolução genĂ´mica de X. laevis ĂŠ uma pouco mais complexa: seu processo de especiação foi decorrente da hibridação (alopoliploidia) de duas espĂŠcies parentais jĂĄ extintas, ambas portadoras de 2n=18 cromossomos, gerando um cariĂłtipo 2n=36 cromossomos. Este evento de alopoliploidia ocorreu a 17-18 milhĂľes de anos atrĂĄs e atualmente ĂŠ possĂvel distinguir dois subgenomas nesta espĂŠcie, uma chamado de subgenoma S (small) e o outro de L (large). Rearranjos intracromossĂ´micos e deleçþes marcaram a reorganização cariotĂpica desta espĂŠcie. Ficamos intrigados em estudar conteĂşdos de DNAs repetitivos, especificamente, dos retrotransposons da ordem DIRs, e entender como ocorreu a evolução desses elementos no genoma dessas duas espĂŠcies, que partilharam de um Ăşltimo ancestral comum a cerca de 48 milhĂľes de anos!
O trabalho foi focado nos retrotransposons da superfamĂlia DIRs-like e Ngaro-like. Avaliamos a diversidade de cĂłpias, sua estrutura e nĂvel de integridade molecular e estimamos temporalmente ondas de amplificação desses elementos em cada genoma. Nossos dados revelaram maior sucesso dos elementos da superfamĂlia DIRs-like em ambos genomas, com maior nĂşmero de cĂłpias e diversidade de famĂlias. Curiosamente, encontramos ondas de invasĂŁo antiga desses elementos no genoma de X. laevis, possivelmente jĂĄ ocorridas nas espĂŠcies parentais antes do processo de hibridação. Avaliando dados de expressĂŁo, identificamos atividade transcricional de cĂłpias de ambas as famĂlias nos genomas, o que foi concordante com a identificação de cĂłpias ainda intactas neste genoma, com todo potencial de realizarem novas inserçþes. Os dados foram recentemente pulicados no periĂłdico internacional G3 (Genes, Genome, Genetics) e integram os estudos realizados no projeto de tese da aluna Camilla Borges Gazolla e conta com a colaboração da Dra. Adriana Ludwig do Instituto Carlos Chagas, especialista em evolução de elementos genĂŠticos mĂłveis.
O prĂłximo passo deste trabalho serĂĄ mapear essas cĂłpias nos cromossomos de X. tropicalis e de Pipa carvalhoi (espĂŠcie do gĂŞnero-irmĂŁo, e tambĂŠm portadora de 2n=20 cromossomos) por meio de ensaios de hibridação “in situ” fluorescente (FISH) e assim demonstrar sua organização nos cariĂłtipos e seu papel na evolução e diversificação cromossĂ´mica nesta famĂlia de anuros.
Quer saber mais sobre esta publicação? acesse o link e boa leitura!
Elementos de transposição (TEs) sĂŁo sequĂŞncias de DNA com capacidade (real ou potencial) de se mobilizar de uma parte do genoma para outra. Os genomas dos vertebrados estĂŁo repletos deste tipo de sequĂŞncias, que compĂľem um elemento adicional para o estudo da variabilidade genĂŠtica. No anfĂbio Anuro Rhinella marina, analisado neste trabalho, os TEs representam cerca de 30% das sequĂŞncias deste genoma! AlĂŠm de serem sequĂŞncias importantes para a evolução dos genomas, evidĂŞncias tem sugerido ativação da atividade transcricional destes elementos frente ĂĄ condiçþes de estresse celular. Alguns trabalhos tem demonstrado que tal atividade transcricional pode ser peça fundamental para a adaptabilidade de organismos a novos ambientes e condiçþes (atuando como sequencias regulatĂłrias de vias biolĂłgicas, por exemplo), o que despertou interesse de nossa equipe em avaliar se existia tal atividade em R. marina quando submetida ĂĄ condiçþes de estresse imunolĂłgico causado artificialmente. Rhinella marina nĂŁo foi uma espĂŠcie escolhida simplesmente ao acaso para condução de tal estudo! Isso porque a R. marina ĂŠ uma importante espĂŠcie invasora, muito bem adaptada ĂĄ ocupação de ambientes climaticamente muito distintos de sua ĂĄrea de ocupação ancestral. Em muitas regiĂľes do mundo, a espĂŠcie chega a ser um importante bioinvasor.
A nossa principal pergunta foi: “Existe expressĂŁo de TEs em indivĂduos de R. marina submetidos Ă condiçþes de estresse? Para isso, analisamos dados de RNA-seq originados de um trabalho previamente publicado por Gardner e colaboradores (2018). Neste trabalho os autores dividiram sua amostra entre animais submetidos Ă estresse imunolĂłgico e outra de animais que nĂŁo foram submetidos Ă estresse (controle). Quando analisamos os dados obtidos por esses autores conseguimos identificar a presença de transcritos diferencialmente expressos entre os grupos tratados e nĂŁo-tratados, indicando que R. marina quando submetida ĂĄ condiçþes de estresse podem reativar a atividade transcricional de TEs como uma das respostas genĂ´micas ĂĄ essa nova condição.
As anĂĄlises foram realizadas em um projeto piloto executado durante a disciplina “Elementos GenĂŠticos MĂłveis”, ministrada pelo prof. Dr. Daniel Pacheco Bruschi no PPG em GenĂŠtica da UFPR e contou com a participação de alunos do LabCECA e de outros laboratĂłrios da UFPR e da Fiocruz.
Esses dados reacendem a discussĂŁo sobre o impacto dos TEs nos genomas de eucariotos e abre perspectivas para futuras investigaçþes sobre o significado biolĂłgico desta expressĂŁo diferencial desses elementos e seu possĂvel papel no rĂĄpido sucesso adaptativo destes organismos ao redor do mundo.
Ficou interessado neste trabalho? Clique aqui e baixe o artigo e boa leitura!!!
O artigo intitulado “Paraphyly in the giant torrent-frogs (Anura: Hylodidae: Megaelosia) and the description of a new genus“, de primeira autoria do Dr. Stenio Eder Vittorazzi, pĂłs-doutorando do LabCECA, combinou dados cromossĂ´micos e de segmentos de genes de DNA mitocondrial e nuclear na descrição de um novo gĂŞnero denominado Phantasmarana. A etiologia do nome vem de uma combinação das palavras latinas phantasma (= fantasma) + rana (=anuros), em referĂŞncia a extraordinĂĄria raridade dos anuros deste gĂŞnero na natureza e devido a enigmĂĄticos sons emitidos por estes anfĂbios, cujo seu significados biolĂłgicos ainda nĂŁo sĂŁo bem compreendidos pelos pesquisadores. As seis espĂŠcies que compĂľem o gĂŞnero Phantasmarana eram historicamente agrupadas dentro do gĂŞnero Megaelosia. No entanto, ĂĄrvores filogenĂŠticas inferidas pelos autores do estudo sugeriram que o atual gĂŞnero Megaelosia ĂŠ monotĂpico (formado por apenas uma Ăşnica espĂŠcies – Megaelosia goeldii) e grupo-irmĂŁo de Hyloides, ambos os gĂŞneros com espĂŠcies portadoras de 2n=26 cromossomos. Phantasmarana ĂŠ grupo-irmĂŁo do arranjo Megaelosia+Hyloides e ĂŠ constituĂdo por espĂŠcies portadoras de 2n=28, 2n=30 e 2n=32. O prĂłximo passo de Vittorazzi e colaboradores ĂŠ investigar a origem desta variação cromossĂ´mica numĂŠrica encontrada em Phantasmarana e assim, compreender melhor os caminhos de diversificação genĂ´mica ao longo da evolução deste grupo tĂŁo interessante.
Devido Ă grande similaridade morfolĂłgica detectada em inĂşmeras espĂŠcies de anfĂbios, dĂşvidas taxonĂ´micas sĂŁo frequentemente relatada por pesquisadores, principalmente quando comparam pares de espĂŠcies baseados apenas em observaçþes de morfologia externa. Para resolver esses tipos de questionamento, pesquisadores tĂŞm recorrido a anĂĄlises integrativas, combinando dados fenotĂpicos (como morfologia interna e externa de adultos e de girinos, parâmetros bioacĂşsticos, por exemplo) com dados genotĂpicos (genĂŠticos e citogenĂŠticos). Entre os dados genotĂpicos, estudos incluindo o uso de sequĂŞncias de DNA (mitocondrial e/ou nuclear) buscam inferir o relacionamento filogenĂŠtico entre as espĂŠcies comparadas. AnĂĄlises citogenĂŠticas, comparam a distribuição de marcadores cromossĂ´micos nos cariĂłtipos das espĂŠcies e tambĂŠm tem sido incluĂda como ferramenta adicional nestes estudos taxonĂ´micos. Para tentar resolver o dilema sobre a taxonomia de duas espĂŠcies, Crossodactylus gaudichaudii and C. aeneus, o pĂłs-doutorando Stenio Eder Vittorazzi analisou fragmentos de genes do DNA mitocondrial (rDNA 12S, 16S e tRNAVal), utilizou metodologias de citogenĂŠtica clĂĄssica e molecular e analisou seus resultados comparativamente com dados fenotĂpicos jĂĄ depositados na literatura.
     O trabalho intitulado âCytogenetic and genetic data support Crossodactylus aeneus MĂźller, 1924 as a new junior synonym of C. gaudichaudii DumĂŠril and Bibron, 1841 (Amphibia, Anura)â, foi recentemente publicado no periĂłdico Genetics and Molecular Biology e apresenta argumentos para a sinonimização das duas espĂŠcies, uma vez que sĂŁo geneticamente indistinguĂveis.  Crossodactylus gaudichaudii and C. aeneus foram indistinguĂveis em anĂĄlises de morfologia externa e interna de adultos, na comparação da morfologia de girinos, em parâmetros bioacĂşsticos, alĂŠm de apresentarem uma sobreposição geogrĂĄfica em sua distribuição na regiĂŁo de Mata Atlântica no sudeste do Brasil. Faltava a cartada final, agora com dados genĂŠticos. Foram empregados testes de delimitação de espĂŠcies, onde as duas espĂŠcies nominais foram recuperadas como uma mesma linhagem evolutiva. AlĂŠm disso, o nĂşmero, morfologia dos cromossomos, posição das regiĂľes organizadoras de nuclĂŠolos (NOR) foram idĂŞnticas em ambos cariĂłtipos. Vittorazzi tambĂŠm mapeou cromossomicamente o DNA satĂŠlite PcP190 (satDNA) e outras duas repetiçþes de microssatĂŠlite ([GA]15 e [CA]15). Assim, por todos estes dados apresentados, as duas espĂŠcies nĂŁo podem ser diferenciadas, o que suporta a hipĂłtese de que correspondem a uma mesma e Ăşnica unidade taxonĂ´mica, sendo portanto C. aeneus um sinĂ´nimo-jĂşnior de C. gaudichaudii.
    Quer conhecer mais sobre este interessante artigo, clique aqui e baixe o artigo. Boa leitura!
Com cerca de 28 espĂŠcies, os anfĂbios do gĂŞnero Cycloramphus possuem muitas lacunas sobre sua histĂłria natural. Parte delas, deve-se dificuldade de encontrar estes espĂŠcimes na natureza por apresentarem uma grande suscetibilidade ao declĂnio populacional, em maior parte devido a ação do homem nos locais em que estas espĂŠcies habitam: Mata atlântica e Floresta das AraucĂĄrias dos estados de Santa Catarina e ParanĂĄ. Estes fatores motivaram a inclusĂŁo de vĂĄrias dessas espĂŠcies no Plano de Açþes Nacionais de Conservação das EspĂŠcies Ameaçadas de Extinção hĂĄ alguns anos.
No esforço de acrescentar informaçþes a respeito deste gĂŞnero de anfĂbios, o trabalho intitulado âCytogenetic characterization and mapping of the repetitive DNAs in Cycloramphus bolitoglossus (Werner, 1897): more clues for the chromosome evolution in the genus Cycloramphus (Anura, Cycloramphidae)â, publicado na revista Plos One, apresenta dados inĂŠditos sobre a espĂŠcie Cycloramphus bolitoglossus, um anfĂbio endĂŞmico da Mata Atlântica no estado do ParanĂĄ. O artigo descreve pela primeira vez o cariĂłtipo dessa espĂŠcie, apresentando o nĂşmero e morfologia dos cromossomos, padrĂŁo de distribuição da heterocromatina constitutiva e posição das RegiĂľes organizadoras de nuclĂŠolo (NORs). Essas sĂŁo caracterĂsticas fundamentais para o conhecimento sobre o complemento cromossĂ´mico das espĂŠcies e ponto de partida para as comparaçþes entre os cariĂłtipos das espĂŠcies-irmĂŁs. Assim, quando comparamos os cariĂłtipos de espĂŠcies relacionadas, ĂŠ possĂvel sugerir hipĂłteses evolutivas com base em rearranjos cromossĂ´micos que fornecem pistas sobre as divergĂŞncias entre as espĂŠcies do gĂŞnero Cycloramphus.
O trabalho foi desenvolvido pela bolsista de PIBIC (Programa Institucional de Bolsas de Iniciação CientĂfica) Gislayne de Paula Bueno, acadĂŞmica do curso de Medicina VeterinĂĄria da Universidade Federal do ParanĂĄ. O projeto envolveu o laboratĂłrio de CitogenĂŠtica Evolutiva e Conservação Animal do Departamento de GenĂŠtica da UFPR e da equipe do LaboratĂłrio de Dinâmicas EcolĂłgicas, do Departamento de Zoologia da UFPR. TambĂŠm integrou a equipe o Dr. Kaleb Pretto Gatto, pĂłs-doutorando da Universidade Estadual Paulista âJĂşlio de Mesquitaâ (Unesp/Rio Claro).
Como grande achado, a equipe descreveu pela primeira vez na famĂlia Cycloramphidae a presença de um DNA satĂŠlite: o PcP190. DNAs satĂŠlites constituem uma classe de DNAs nĂŁo-codificantes (que nĂŁo transcrevem para nenhum produto celular aparentemente) e que estĂŁo presentes repetidos centenas de cĂłpias em uma mesma regiĂŁo do genoma (repetidos âin tandemâ). Esses DNAs repetitivos constituem grande fração dos genomas de eucariotos e sĂŁo importantes componentes estruturais dos cromossomos e muitas evidĂŞncias sugerem sua importância como substrato durante a evolução cromossĂ´mica das espĂŠcies. Neste trabalho, os autores encontraram grande similaridade estrutural com cĂłpias do DNA ribossomal 5S (rDNA 5S), confirmando o mesmo padrĂŁo jĂĄ relatado em outros Anuros. O trabalho tambĂŠm localizou em quais cromossomos do cariĂłtipo esse DNA satĂŠlite estava presente, utilizando para isso a tĂŠcnica de hibridação âin situâ fluorescente (FISH). Acredita-se que o DNA satĂŠlite PcP190 tenha se originado de um processo de duplicação do gene de rDNA 5S e tenha acumulado mutaçþes ao longo do tempo que resultaram no surgimento dessa nova famĂlia de DNA satĂŠlite, aparentemente restrita Ă algumas poucas linhagens de Anuros.
Esses novos dados adicionam um nova pĂĄgina a histĂłria do gĂŞnero Cycloramphus e no estudo desse DNA satĂŠlite em Anuros e abre novas perspectivas para a compreensĂŁo o papel desses DNAs nos genomas desse grupo.
https://doi.org/10.1371/journal.pone.0245128
Hoje foi publicado, no European Journal of Taxonomy, um artigo cientĂfico que descreve uma belĂssima nova espĂŠcie brasileira. Uma singela homenagem Ă cultura nordestina e ao rei do baiĂŁo, Luiz Gonzaga, um dos âarquitetos da mĂşsica brasileiraâ.
O professor Daniel Pacheco Bruschi, do Programa de PĂłs-graduação em GenĂŠtica, liderou o estudo, que conta tambĂŠm com a participação do doutorando Johnny Sousa Ferreira e dos pesquisadores Dr. LuĂs Felipe Toledo e Felipe Silva de Andrade da Unicamp e Isabelle Aquemi Haga da Universidade Federal de Uberlândia. O trabalho foi financiado pela Fundação de Amparo Ă Pesquisa do Estado de SĂŁo Paulo (FAPESP) e pelo Conselho Nacional de Desenvolvimento CientĂfico e TecnolĂłgico (CNPq).
Como vocês jå podem desconfiar, esse bicho ocorre lå no Nordeste, nos estados que ficam ao norte do grandioso Rio São Francisco (RSF), o velho Chico. Este importante rio teve um papel central na história evolutiva da nova espÊcie, bem como da sua espÊcie-irmã, o P. nordestinus. Alteraçþes no seu curso hå milhares de anos causaram um isolamento geogråfico entre as populaçþes ancestrais. Isso não Ê muito bacana? Nós chamamos isso de evento vicariante, que Ê quando uma barreira geogråfica isola populaçþes que antes estavam conectadas, originando, após milhares de anos, duas espÊcies distintas. Portanto, nós observamos atualmente o P. gonzagai ao norte do RSF e o P. nordestinus ao sul do RSF. O rio atua como uma barreira geogråfica que isola reprodutivamente ambas espÊcies.
Entretanto, sabemos que jå estå em curso a Transposição do Rio São Francisco, uma grande obra que direciona a ågua do RSF para regiþes nordestinas que sofrem bastante com as secas. Qual serå o impacto ambiental dessa obra sobre o P. gonzagai? Temos nossas predileçþes e indicamos caminhos para se acompanhar esse impacto lå no artigo. De qualquer forma, devemos ficar em alerta sobre essa situação.
Logo abaixo estå um videoclipe da música que a banda Papo de Sapo gravou sobre a nossa descoberta! A pessoa que estå cantando Ê o Daniel Gonzaga, NETO do Gonzagão e FILHO do Gonzaguinha!!! Ele topou essa parceria e ficou muito bacana. Pessoal, arraste os móveis da sua sala e bora dançar esse baião maravilhoso. Ajude-nos na divulgação! Compartilhe nas redes sociais e grupos de WhatsApp. Viva a biodiversidade e a música brasileiras!!!
Link do artigo cientĂfico: https://europeanjournaloftaxonomy.eu/index.php/ejt/article/view/1147
#brazilianbiodiversity #amphibians #frogs #nordeste #riosaofrancisco #especienova #brazil #fauna
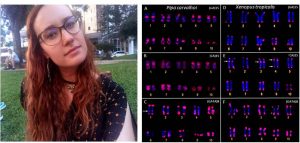
Pipa carvalhoi Ê um anfĂbio que habita regiĂľes de Mata Atlântica e ĂĄreas sob influĂŞncia da Caatinga no Nordeste do Brasil. JĂĄÂ Xenopus tropicalis ocupa principalmente a regiĂŁo da Ăfrica subsariana. Em comum, as duas espĂŠcies pertencem a famĂlia Pipidae, que reĂşne espĂŠcies de anfĂbios que sĂŁo essencialmente aquĂĄticos. Estima-se que esses gĂŞneros tenham partilhado de um Ăşltimo ancestral comum a pelo menos 136 milhĂľes de anos, sendo a separação do antigo continente Godwanna a principal hipĂłtese para essa diversificação. Em nosso Lab, temos buscado investigar marcas deixadas no genoma dessas espĂŠcies que possam nos ajudar a recontar sua histĂłria de diversificação. Essas duas espĂŠcies apresentam mesmo nĂşmero diploide de cromossomos (2n=20) e compartilham de semelhanças na morfologia de muitos pares de seus cariĂłtipos, quando apenas estudados por tĂŠcnicas de citogenĂŠtica clĂĄssica.
O artigo “Evolutionary dynamics of the repetitive DNA in the karyotypes of Pipa carvalhoi and Xenopus tropicalis (Anura, Pipidae)”, recentemente publicado na revista Frontiers in Genetics analisa a histĂłria evolutiva das duas espĂŠcies atravĂŠs de seus cariĂłtipos. Mais especificamente, sequĂŞncias de DNA repetitivas (motivos microssatĂŠlites) foram mapeadas nos cariĂłtipos das duas espĂŠcies e revelaram padrĂľes de distribuição dessas sequencias bastante discrepante entre os cromossomos. Mais que isso, os resultados obtidos permitiram a identificação de rearranjos cromossĂ´micos ocorridos ao longo da evolução dessas duas espĂŠcies. O trabalho foi originado da dissertação de mestrado de Michelle Louise Zattera, do programa de PĂłs Graduação em GenĂŠtica da UFPR (PPG-GEN). Esses novos dados contribuem para uma melhor compreensĂŁo da evolução cromossĂ´mica em Pipidae e reforçam o papel dos DNAs repetitivos na remodelação dos cariĂłtipos ao longo da evolução e de sua importância para a estrutura e funcionamento dos genomas. Ficou interessado? Visite em nossa pĂĄgina Publicaçþes e fique por dentro desse artigo!
